Nytt genotypedatasett i produksjon
Genotype-dataene som blir brukt i avlsverdiberegningene har nå blitt forbedret, og man vil ved beregningene i sommer kunne ha opplevd en litt større endring i avlsverdiene enn det man har mellom vanlige kjøringer.
arne.gjuvsland@geno.no
Avlsforsker i Geno
oyvind.nordbo@geno.no
Avlsforsker i Geno
havard.melbo.tajet@geno.no
Avlssjef i Geno
Geno anvendes all tilgjengelig informasjon, både stamtavle, genotyper og dataregistreringer inn i en stor statistisk modell for beregning av avlsverdier. For å få en så riktig rangering av dyr som mulig, er det viktig at genotypedata og stamtavledata er harmonisert. Vi hadde en større oppretting og oppgradering av genotypedataene for ca. to år siden som var omtalt i Buskap nr. 6 i 2018 (se buskap.no), og vi får nå en mindre oppgradering for å bedre presisjonen ytterligere. Utslagene nå vil være mye mindre enn for oppgraderingen for to år siden, men man vil som resultat av forbedringen kunne oppleve at dyr vil kunne endre avlsverdi med opptil 5 indekspoeng. Figuren viser at omkring 90 prosent av dyra ikke endrer seg mer enn +/- 2 indekspoeng.

Genotype-dataene som blir brukt i avlsverdiberegningene blir stadig bedre, og med den siste oppgraderingen vil forskjellen i avlsverdi mellom ikke-genotypa og genotypa dyr være helt borte. Foto: Jumpstory
Fire nye/forbedrete elementer i oppgraderingen
Nytt referansegenom: Imputeringa (innfylling av manglende eller feil genotypedata) er nå basert på det nye referansegenomet for storfe. Arbeidet med nytt referansegenom var omtalt i Buskap nr. 4 i 2020 (se buskap.no) og handler om at kartet for kuas arvemateriale er blitt forbedret. Dette gjør at det blir færre feil i de genotypene som går inn i avlsberegninga.
Oppdatert programvare: Dataprogrammet FImpute, som vi bruker for å fylle inn manglende eller feil genotyper (imputering) har kommet i ny versjon. Dette reduserer også mengden feil i genotypene.
Nytt SNP-sett: Utvalget av genetiske markører som blir brukt inn i avlsverdiberegninga er forbedret. Antallet markører (SNP-er) er omtrent det samme, økning fra 109 000 til 122 000, men vi har nå vært enda strengere med filtreringa og fjernet markører som viser mange avvik fra mendelsk nedarving. I tillegg har vi lagt til noen nye markører som har god kvalitet.
-
Forbedringer i innstillingene for utregninger med genomiske slektskap.
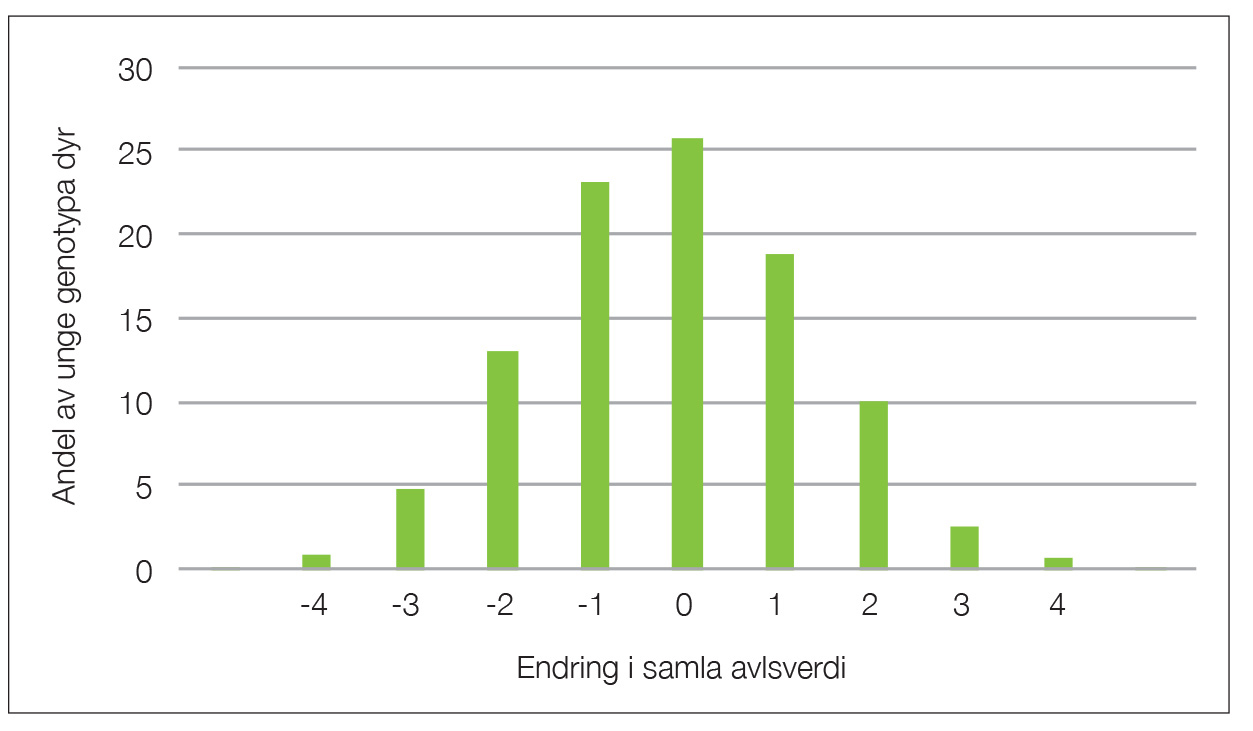
Figur. Endring i samla avlsverdi
Bedre seleksjon – økt avlsframgang
Den viktigste konsekvensen vil være at systematiske forskjeller i avlsverdiene mellom genotypa og ikke-genotypa dyr forsvinner. Før denne endringa gikk dyr i gjennomsnitt opp i underkant av ett poeng i den samla avlsverdien når de ble genotypa. Nå vil denne forskjellen i avlsverdi mellom ikke-genotypa og genotypa dyr være helt borte. Dette vil gjøre at man kan gjennomføre en bedre seleksjon og vil derfor øke avlsframgangen til NRF.

