Riktigere slektskap med genotyper
Genomisk slektskap er et mål på hvor stor andel av genene dyr har til felles og er et riktigere mål enn det man har utnyttet i tradisjonell avl.
Kromosom
Storfegenomet består av 30 kromosompar
Hvert kromosompar består av to kromosomer, der dyret har arvet et kromosom fra far og et fra mor
Et kromosom består av to DNA-tråder som er koblet sammen av to ulike basepar
-
Det er fire baser som utgjør de to baseparene:
A og T (adenin og tymin)
C og G (cytosine og guanine)
Et gen er et eller flere basepar som koder for en eller flere egenskaper
SNP – single nucleotide polymorphism – er et punkt på DNA’et der det finnes variasjon i basepar mellom individer. De er jevnt fordelt utover DNA, og de er funnet til stort sett å ligge i nærheten av et gen som koder for en egenskap og arves dermed sammen med genet. SNP kalles derfor også for markører.
Ved å erstatte et teoretisk slektskap basert på stamtavle med et målt genomisk slektskap, blir avlsverdiene sikrere.
Stamtavla
Før genotypeinformasjon var tilgjengelig var et slektskap basert på stamtavla det beste målet på genetisk likhet og slektskap. Det ble beregnet et slektskap til hvert enkelt dyr som sa noe om hvor mye dyra var i slekt og hvor mange felles nedarvede gener de ulike dyrene hadde.
Slektskap til mor og far
Et dyr er alltid 50 prosent i slekt med sin mor og 50 prosent i slekt med sin far. Et avkom arver halvparten av genene fra far og halvparten av genene fra mor (se bilde 1).

Bilde 1: Oksekalven har arvet 50 prosent av genene sine fra far og 50 prosent av genene sine fra mor.
Overkrysning av kromosomer
Som bilde 1 illustrerer, kan det hos både mor og far skje en overkrysning mellom kromosomene. For eksempel er kromosomet (i blått) oksekalven har fått fra sin far en kombinasjon av far sine to kromosomer. Bilde 2 illustreres en slik overkrysning.

Bilde 2: Overkrysning mellom to kromosomer. Denne overkrysningen kan skje på ulike steder på kromosomene. Posisjon for en eventuell overkrysning kan variere. De samme foreldrene kan gi et annet avkom med helt andre genkombinasjoner, slik som bilde 3 illustrerer. Denne oksekalven er også 50 prosent i slekt med sin far og 50 prosent i slekt med sin mor, og det har skjedd en overkrysning av kromosomene på et annet sted enn for oksekalven i bilde 1.
Slektskap mellom helsøsken
De to oksekalvene på bilde 1 og bilde 3 er begge 50 prosent i slekt med hver av sine foreldre, men hvor mye er de i slekt med hverandre? Tidligere ville vi antatt at de var 50 prosent i slekt med hverandre. Det vil si at de har arvet 25 prosent av de samme genene fra far og 25 prosent av de samme genene fra mor. Men som vi kan se illustrert i bilde 4, er ikke dette helt presist. Fordi det har skjedd en overkrysning på ulike posisjoner på kromosomene hos både mor og far, har kalvene arvet ulike kromosomsegmenter. Når man sammenligner genotypene til to søsken, kan man se at noen segmenter er identiske mens andre er ulike. Det genomiske slektskapet mellom dyra er et mål på hvor stor andel av genene som er identiske. Teoretisk kan helsøsken være alt fra 0 prosent til 100 prosent i slekt, men vanligvis varierer dette fra 40 prosent til 60 prosent.

Bilde 3: En oksekalv har arvet 50 prosent av genene sine fra far og 50 prosent av genene sine fra mor, men har arvet ulike gener enn sin helbror på bilde 1.

Bilde 4: Oksekalver som er helsøsken trenger nødvendigvis ikke være 50 prosent i slekt (kalven fra bilde 1 til høyre og kalven fra bilde 3 til venstre)
Hva har det å si for avlsverdien til en kalv?
Et mer presist slektskapsmål gjør at avlsverdien til dyret blir sikrere beregnet. Ved å genotype dyra og finne ut hvilke genmateriale de har til felles, kan vi med større sikkerhet vite om avkommet faktisk har arvet de gunstige genene som er knyttet til egenskaper vi er interessert i. Dette kan illustreres med et eksempel om oksekalven Filliokus som er etter eliteoksen Braut (bilde 5 og 6). Filliokus har fire eldre halvsøstre som er i sin første laktasjon. Han har ikke en egen fenotype eller døtre med fenotyper, så avlsverdien hans baserer seg på andre slektningers prestasjoner.
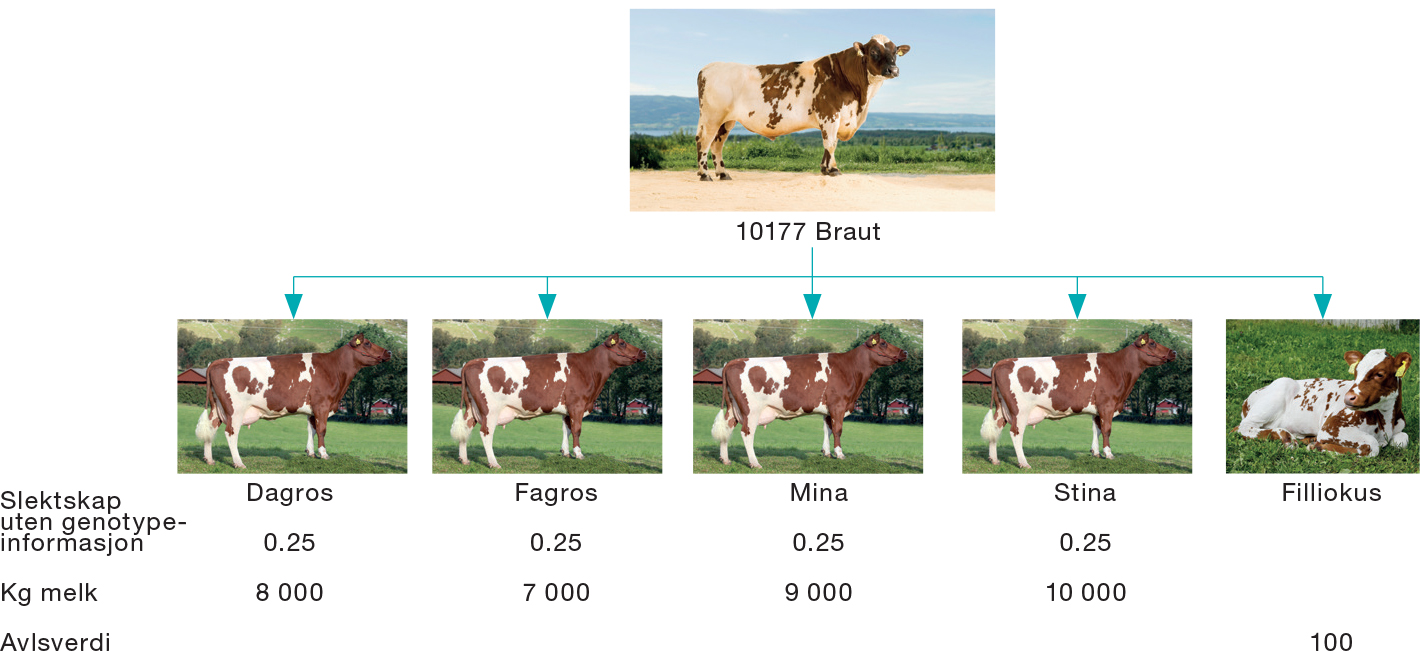
Bilde 5: Illustrasjon på slektskap mellom dyr ved bruk av stamtavleinformasjon, og hvordan det påvirker avlsverdien.
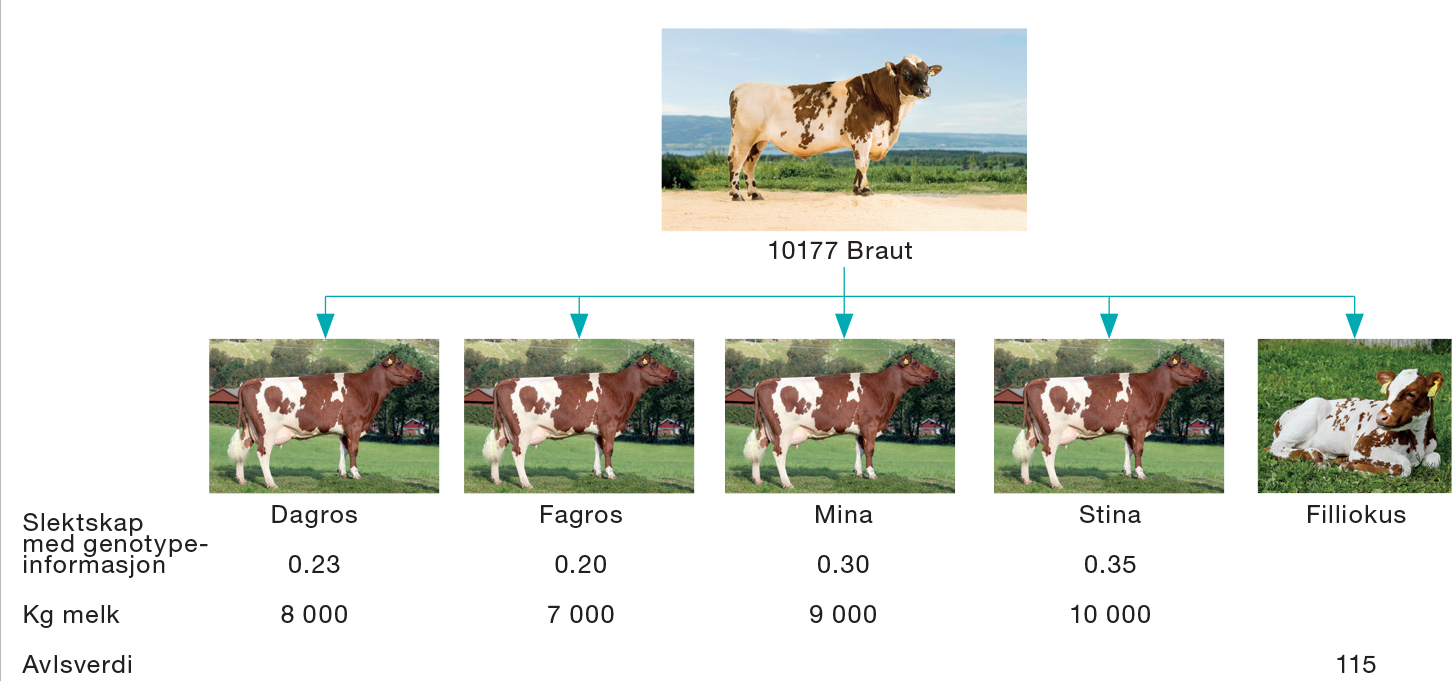
Bilde 6: Illustrasjon på ulik genomisk slektskap og hvordan det kan påvirke avlsverdien til oksekalven.
Uten genotypeinformasjon
Med tradisjonelt avlsarbeid er det informasjon fra stamtavla som avgjør slektskapet mellom dyr. Bilde 5 viser at slektskapet mellom Filliokus og halvsøstrene er 25 prosent for alle. For å beregne avlsverdien til et dyr, brukes slektskap basert på stamtavla for å vekte fenotypemålingene av slektninger inn i avlsverdien. I dette eksempelet betyr det at fenotypene til halvsøstrene til Filliokus vil bidra like mye inn i beregningen av avlsverdien til Filliokus. Det vil si at fenotypen til alle halvsøstre vektes med 0,25 inn i avlsverdien til Filliokus, og basert på dette får Filliokus en avlsverdi for kg melk på 100.
Med genotypeinformasjon
Hvis både far Braut, Filliokus og de fire halvsøstrene: Dagros, Fagros, Mina og Stina er genotypet, kan et genomisk slektskap beregnes mellom dem. Dette gjør at vi kan tallfeste likheten mellom genotypene til to og to dyr. Bilde 6 illustrere dette. Her kan vi se at slektskapet mellom Filliokus og de fire halvsøstrene varierer mellom 23 prosent og 35 prosent. Ved å benytte det genomiske slektskapet får man en mer riktig vekting av slektningers fenotype inn i avlsverdien. Dette medfører at avlsverdien til kalven vil kunne gå opp eller ned, alt ettersom hvilke dyr den får økt eller redusert slektskap til. Fordi Filliokus er mer i slekt med Mina og Stina, som også melker mer, vil Filliokus sannsynligvis ha flere av de gunstige genene for melk til felles med Mina og Stina. Avlsverdien vil derfor øke.

