Høge avlsverd ier på NRF
Mange opplever at de har svært høge avlsverdier på NRF-dyra sine. Spesielt merkes dette nå når et betydelig antall hunndyr, både kviger og eldre kuer har blitt genotypa. Det er flere årsaker til dette.
Leder avdeling for for FoU og implementering
havard.melbo.tajet@geno.no

Med GS har avlsframgangen skutt fart. Større spredning av indekser og at det tar tid før skaleringsbasen er i takt med avlsframgangen gjør at de beste dyra får for høge avlsverdier. Sikkerheten på indeksene er god, men det er altså nivået som er i høyeste laget.
Foto: Rasmus Lang-Ree
Den fullstendige overgangen til GS (genomisk seleksjon) i februar 2016, ga oss muligheten til å velge eliteokser blant ferdige avkomsgranska, halvferdige og unge okser som ikke var kommet ordentlig i gang enda, tilsammen omkring 400 okser. Aldri før hadde vi kunnet selektere blant så mange okser. Indeksnivået på eliteoksene fikk et fantastisk løft. Dette har gitt stor avlsframgang som vi nå ser i form av svært høgt indeksnivå på kviger og oksekalver.
30 000 genotypa NRF-dyr
Tidligere var kuindeksene i stor grad basert på gjennomsnitt av mor og far, og det var kun kuas egen prestasjon med moderat til lav arvbarhet som kunne skille henne fra dette gjennomsnittet. Sikkerheten var ganske lav og dermed også spredningen på kuenes indekser. Nå har svært mange produsenter begynt genotyping av hunndyra sine. For et år siden hadde vi ca. 18000 genotyper i GS-beregningene. I skrivende stund (12. januar 2018) har vi 30 479 genotypa dyr i beregningene og med langt større andel hunndyr. Disse har betydelig større sikkerhet og spredninga øker. Det vil si at vi nå oppdager noen svært gode dyr som tidligere hadde middels indeksnivå. De får nå vist hvor gode de faktisk er. Her dukker det altså opp en del ekstrem-dyr. Tilsvarende er det også noen dyr som viser seg å være dårligere enn tidligere antatt.
Lavere middel, men større spredning
Tradisjonelt har vi nedskalert indeksene ved hver avkomsgransking med påfølgende eliteokseutvalg. Dette er for å unngå at det går «inflasjon» i indeksene. Denne nedskaleringa (eller «devalueringa») har skjedd i takt med avlsframgangen. Nullpunktet defineres løpende av det vi kaller skaleringsbasen. Der inngår de siste avkomsgranska oksene. I desember 2017 besto basen av okser som fikk avkomsgrupper fra og med 2014 til og med 2016. Denne rullerende skaleringsbasen har ikke rukket å bli påvirket av den kraftige avlsframgangen enda. Dette er en vesentlig årsak til at de beste dyra i dag har betydelig høgere avlsverdier enn de beste dyra ved innføring av GS. Ved siste endring av skaleringsbasen gikk nullpunktet ned med ett indekspoeng. Samtidig økte spredninga på indeksene noe, slik at dyr med indeks på ca. 30 ikke fikk noen endring og dyr med over 50 steg noe. Det betyr at dyra genetisk sett hverken er bedre eller dårligere enn før, men at skalaen er endra med lavere middel, men litt større spredning. Vi jobber nå med å gjøre om skaleringsbasen til en kubase som er stor nok til at spredninga blir så stabil som mulig. Basen må være stor nok til å oppnå stabilitet samtidig som den må være ung nok til å fange opp taktskifter i avlsframgangen så tidlig som mulig.
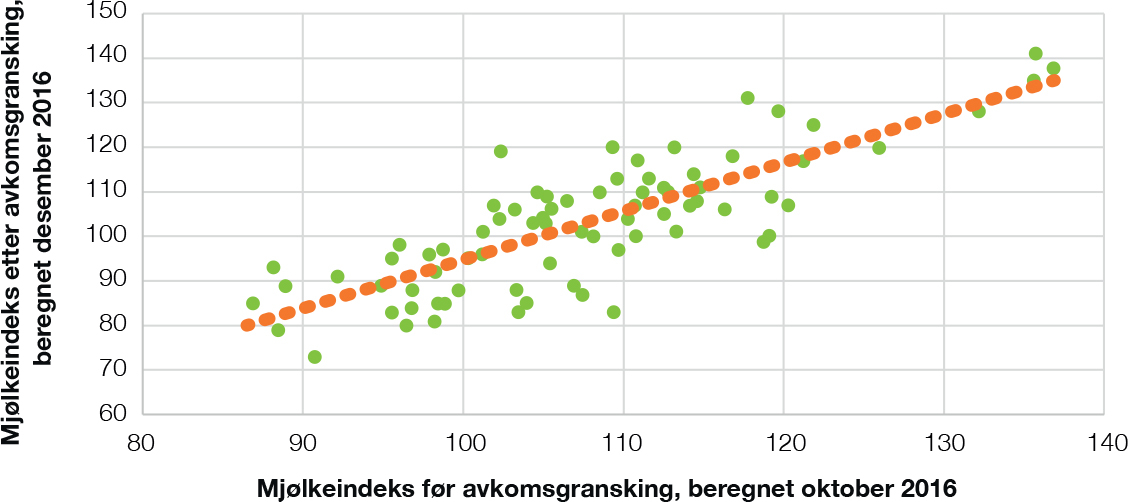
Figur. Sammenhengen mellom mjølkeindeks basert på GS og avkomsgransking.
Hvorfor stiger ku-indeksen etter genotyping?
Så er det utfordringene med at indeksene blir «for høye». Vi har, som tidligere nevnt, sett at totalindeksen til en uselektert genotypa avkomsgruppe er systematisk noe høgere enn gjennomsnittet til foreldrene. Dette er en systematisk feil som kalles bias. Vi har oppdaget at hovedproblemet er knyttet til melkeindeksen. Dette er den egenskapen som har hatt størst genetisk framgang. Vi ser det ved at indeksen til kuene stiger noe når de blir genotypa. Ideelt sett skal indeksen være slik at like mange stiger og synker. Fenomenet er en kjent problemstilling med GS over hele verden, og det jobbes både internt i Geno og internasjonalt med å forstå og å løse dette problemet. Vi har i løpet av november og desember funnet ut at vi kan redusere noe av denne systematiske feilen ved å gjøre følgende tiltak:
Innføre genetiske grupper i våre «single step» GS-avlsverdiberegninger. Dette reduserer den systematiske feilen med ca. 30 prosent.
Innføring av alternativ metodikk for å generere genomiske slektskapskoeffisienter, i kombinasjon med å kutte gamle data.
Eliminere resten av den systematiske feilen etter at avlsverdiberegningsmodellene er ferdig ved å «krympe» spredninga på genotypa dyr uten avkomsgrupper.
Vi har nå begynt å forberede implementering av disse tre punktene. Endring av skaleringsbasen vil komme i samme omgang, og vi forventer at nivået på indeksen blir betydelig lavere med den nye skaleringa. Dette betyr ikke at dyra er dårligere, bare at skalaen er annerledes.
Sikkerhet på indeksene er god
Til tross for behovet for disse justeringene er vi trygge på at sikkerheten på indeksen er god. Vi foretar jevnlig det vi kaller kryssvalidering av GS-indeksene. Kryssvalidering er å stikke fingeren i jorda og sjekke om teorien fungerer i praksis. Det kan gjøres ved at vi ser på sammenhengen mellom GS-indeksene til okser før de får avkom og hvordan døtrene presterer når de kommer i produksjon. Det er ingen prinsipiell forskjell på GS-indeksen til ei kvige og en oksekalv, slik at disse resultatene vil også ha relevans for sikkerhet indeksene til kviger. Sikkerhetene for mange av egenskapene (mjølk, jureksteriør, celletall) ligger mellom 0,6 og 0,7.
God sammenheng mjølkeindeks med og uten døtre
For å illustrere hvordan dette kan se ut har vi laget et plot med mjølkeindekser på okser uten døtre fra oktober 2016 på x-aksen mot mjølkeindeksen etter at oksene har fått 100 til 200 døtre nå i desember 2017 (se figur). Sammenhengen i dette tilfellet ble 0,81, noe som viser at indeksene som ble beregna før oksene fikk døtre stemmer nokså godt overens med indeksene som ble beregna i desember 2017, da vi i tillegg hadde informasjon om dattergruppene. Skalaene har endret seg noe fra oktober 2016 til desember 2018 på grunn av avlsframgang.
Sammenligning på tvers av land i Norden
Det er ofte det spørres om sammenligninger mellom Geno og Viking sine indekser. Det er viktig å få fram at disse ikke er sammenlignbare. Vi har definert vår egen skala og de har definert sin. Dette har vi gjort gjennom hver våre definisjoner av skaleringsbaser. I tillegg er beregningene basert på ulike GS-metodikker, statistiske modeller og for noen egenskaper ulike definisjoner. Når det gjelder totalindeksen er også vår avlsverdi (TMI) bestemt av andre vekter enn deres NTM-avlsverdi. Mange av Viking sine okser er dårlig representert med slektninger i Norge, og våre okser har knapt vært brukt i våre nordiske naboland de seinere år. Dette gjør at oksene kan ha ulike rangeringer i de ulike landene.
Utfordring med RDM-okser
Hvis man skal sammenligne oksers avlsverdier, må det gjøres med indekser beregnet i samme avlsselskap. Videre må man vite om det finnes mange slektninger til den aktuelle oksen med data og genotyper i det landet der indeksen er beregnet. I Norge har vi størst utfordring med RDM-okser (Rød Dansk) der vi har hatt mindre historisk utveksling enn det vi har hatt med SRB (Svensk Rød) og FAY (Finsk Airshire). De kan få noe hjelp av genotyping, men de av RDM-oksene som har mer av Brown Swiss, Holstein og Montbeliarde, har en helt annen kobling mellom DNA-markører og genene som påvirker egenskapene. GS vil derfor fungere dårligere for disse oksene. For okser med mer enn 90 prosent Ayrshire, slik vi har sett i SRB og FAY, vil genotypinga gi bedre mening inn i GS-modellene og sikkerhetene på norske indekser for avkom etter slike importokser blir høyere.
.png)
.png)